Profilage Pharmacologique In Silico
Contexte: Le thème de l’équipe “IsPP” concerne le profilage de petits composés, la caractérisation structurale des cibles thérapeutiques et de leurs régions d’interactions ; la prédiction des interactions composé-cible. Les questions clés sont de caractériser et de prédire d’une part les interactions multiples (polypharmacologie) entre les candidats médicaments et les cibles thérapeutiques, et d’autre part, l’impact des petites molécules à différents niveaux de complexité biologique, conduisant globalement à des effets bénéfiques ou néfastes. Des protocoles in silico sont développés pour l’identification rationnelle de candidats médicaments, combinant l’intelligence artificielle (IA), la chemoinformatique, la bioinformatique et permettant d’intégrer les volumes croissant de données chémo-biologiques, en termes de volume, flexibilité, diversité et sources.
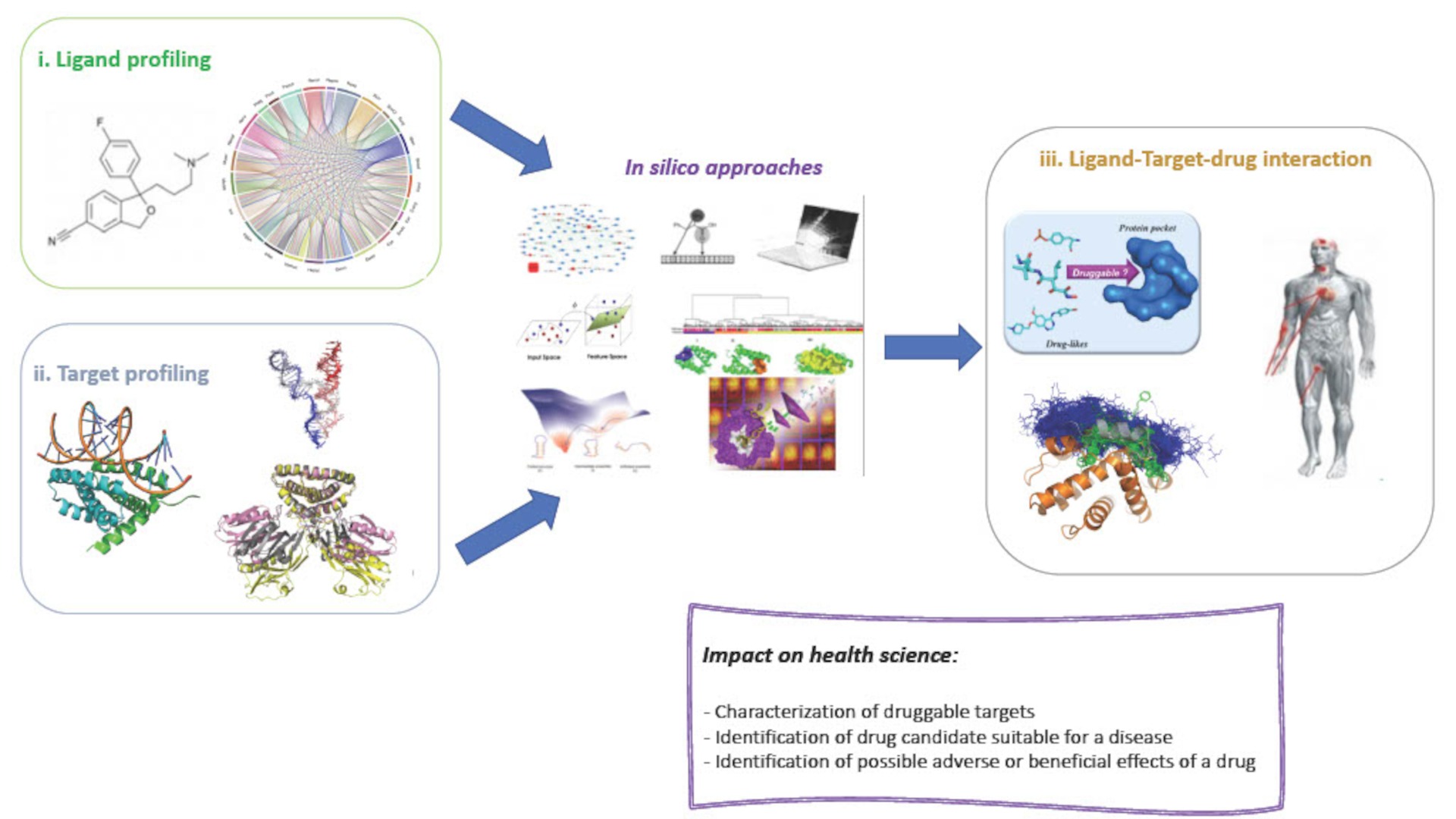
Image principale IsPP
© BFA, UMR 8251 CNRS – Université Paris Cité
Projet : L’objectif de l’équipe est d’explorer le potentiel thérapeutique des cibles et de contribuer à la conception de nouveaux médicaments à l’aide d’outils computationnels. Nous poursuivrons le développement du profilage pharmacologique in silico en appui sur trois axes complémentaires : i) le profilage des ligands intégré à différents niveaux et étendu aux relations ligand-phénotype, ii) l’analyse structurale et de la flexibilité des cibles thérapeutiques (protéiques et d’ARN) et de leurs régions d’interactions et sites de liaison, iii) la prédiction des interactions multiples ligand-cible.
Notre équipe aborde ces questions d’un point de vue informatique en utilisant et en développant des méthodes et des protocoles, sur la base de nos expertises en biostatistique, apprentissage automatique, bioinformatique structurale, simulation de modélisation moléculaire, chémoinformatique, docking et criblage virtuel, biophysique, biologie des réseaux, bioinformatique génomique et biologie chimique computationnelle.
Grâce à l’analyse de l’effet d’une molécule à différents niveaux biologiques sur la santé humaine et au niveau de sa structure, nous espérons enrichir le profilage pharmacologique des petites molécules et guider la conception d’un médicament adapté à une maladie. En proposant une caractérisation détaillée des sites de liaison et de leur flexibilité, nous espérons améliorer la prédiction des partenaires médicamenteux et les résultats de l’amarrage. Nous enrichirons nos approches et explorerons leur pertinence par le biais d’applications expérimentales.
Ces applications seront réalisées à travers des collaborations fortes avec des équipes expérimentales au sein de BFA sur des thématiques des maladies métaboliques (diabète de type 2, obésité), de troubles de la fertilité ; de pathologies neurodégénératives ; mais aussi au niveau de l’Université Paris Cité, au niveau national et international sur une variété de maladies (voire l’onglet projet) et dans le domaine de la sécurité des médicaments et de l’évaluation des risques chimiques.
Ressources et positionnement : L’équipe comprend neuf enseignants-chercheurs et un assistant ingénieur. Elle s’implique fortement dans plusieurs réseaux nationaux ou internationaux aussi bien recherche que pour développer des formations innovantes (bioinformatique, chemoinformatique et drug design). L’équipe s’appuie sur la plateforme de bioinformatique structurale RPBS.
Axes thématiques
Axe 1: Profilage des Ligands
- Basé sur l’intégration des données et la génomique
- Basé sur la structure
Axe 2: Profilage des cibles thérapeutiques
- Cible protéique ou cible RNA
- Caractérisation des régions d’interaction (sites de liaison) en terme de flexibilité et/ou de druggabilité
Axe 3: Profilage pharmacologique
- Prédiction des Interactions multiples
- Prédiction des effets secondaires
Publications récentes
- Gómez-Sacristán P, Simeon S, Tran-Nguyen VK, Patil S, Ballester PJ. Inactive-enriched machine-learning models exploiting patent data improve structure-based virtual screening for PDL1 dimerizers. J Adv Res. 2025 Jan;67:185-196. doi: 10.1016/j.jare.2024.01.024. Epub 2024 Jan 26. PMID: 38280715; PMCID: PMC11725107.
- Granados A, Zamperoni M, Rapone R, Moulin M, Boyarchuk E, Bouyioukos C, Del Maestro L, Joliot V, Negroni E, Mohamed M, Piquet S, Bigot A, Le Grand F, Albini S, Ait-Si-Ali S. SETDB1 modulates the TGFβ response in Duchenne muscular dystrophy myotubes. Sci Adv. 2024 May 3;10(18):eadj8042. doi: 10.1126/sciadv.adj8042. Epub 2024 May 1. PMID: 38691608; PMCID: PMC11062573.
-
Skhoun H, El Fessikh M, Khattab M, Mchich B, Agadr A, Abilkassem R, Dakka N, Flatters D, Camproux AC, Ouzzif Z, El Baghdadi J. A Novel NRAS Variant Near the Splice Junction in Moroccan Childhood Acute Lymphoblastic Leukemia: A Molecular Dynamics Study. Biochem Genet. 2024 Nov 8. doi: 10.1007/s10528-024-10968-2. Epub ahead of print. PMID: 3951408.
-
Yi W, Tran-Nguyen VK, Boumendjel A. One-step synthesis of diaryloxadiazoles as potent inhibitors of BCRP. Future Med Chem. 2024;16(8):723-735. doi: 10.4155/fmc-2023-0322.
-
Caba K, Tran-Nguyen VK, Rahman T, Ballester PJ. Comprehensive machine learning boosts structure-based virtual screening for PARP1 inhibitors. bioRxiv. 2024 Mar 15. doi: 10.1101/2024.03.15.585277.
-
Tariq F, Ahrens L, Alygizakis NA, Audouze K, Benfenati E, Carvalho PN, Chelcea I, Karakitsios S, Karakoltzidis A, Kumar V, Mora Lagares L, Sarigiannis D, Selvestrel G, Taboureau O, Vorkamp K, Andersson PL. Computational Tools to Facilitate Early Warning of New Emerging Risk Chemicals. Toxics. 2024 Oct 12;12(10):736. doi: 10.3390/toxics12100736. PMID: 39453156; PMCID: PMC11511557.
-
Tran-Nguyen, V.-K., Camproux, A.-C. and Taboureau, O. (2024), ClassyPose: A Machine-Learning Classification Model for Ligand Pose Selection Applied to Virtual Screening in Drug Discovery. Adv. Intell. Syst., 6: 2400238. https://doi.org/10.1002/aisy.202400238
-
Ollitrault G, Marzo M, Roncaglioni A, Benfenati E, Mombelli E, Taboureau O. Prediction of endocrine-disrupting chemicals related to estrogen, androgen, and thyroid hormone (EAT) modalities using transcriptomics data and machine learning. Toxics. 2024;12(8):541. doi: 10.3390/toxics12080541.
-
Ollitrault G, Achebouche R, Dreux A, Murail S, Audouze K, Tromelin A, Taboureau O. Pred-O3, a web server to predict molecules, olfactory receptors and odor relationships. Nucleic Acids Res. 2024;52(W1):W507-W512. doi: 10.1093/nar/gkae305.
-
Mattiotti G, Micheloni M, Petrolli L, Rovigatti L, Tubiana L, Pasquali S, Potestio R. Molecular dynamics characterization of the free and encapsidated RNA2 of CCMV with the oxRNA model. Macromol Rapid Commun. 2024;45:2400639. doi: 10.1002/marc.202400639.
- Reboul E, El Hamaoui D, Pasquali S, Gaussem P, Rossi E, Taly A. Molecular modeling of Proteinase-Activated Receptor 1 in complex with Thrombin Receptor Activator Peptide 6. bioRxiv. 2024 Jun 5. doi: 10.1101/2024.06.05.597686.
-
Tubiana, Luca; Alexander, Gareth P; Barbensi, Agnese; Buck, Dorothy; Cartwright, Julyan HE; Chwastyk, Mateusz; Cieplak, Marek; Coluzza, Ivan; Čopar, Simon; Craik, David J; Di_Stefano, Marco; Everaers, Ralf; Faísca, Patrícia FN; Ferrari, Franco; Giacometti, Achille; Goundaroulis, Dimos; Haglund, Ellinor; Hou, Ya-Ming; Ilieva, Nevena; Jackson, Sophie E ; Japaridze, Aleksandre; Kaplan, Noam; Klotz, Alexander R; Li, Hongbin; Likos, Christos N; Locatelli, Emanuele; López-León, Teresa; Machon, Thomas; Micheletti, Cristian; Michieletto, Davide; Niemi, Antti; Niemyska, Wanda; Niewieczerzal, Szymon; Nitti, Francesco; Orlandini, Enzo; Pasquali, Samuela; Perlinska, Agata P; Podgornik, Rudolf; Potestio, Raffaello; Pugno, Nicola M; Ravnik, Miha; Ricca, Renzo; Rohwer, Christian M; Rosa, Angelo; Smrek, Jan; Souslov, Anton; Stasiak, Andrzej; Steer, Danièle; Sułkowska, Joanna; Sułkowski, Piotr; Sumners, De_Witt L; Svaneborg, Carsten; Szymczak, Piotr; Tarenzi, Thomas; Travasso, Rui; Virnau, Peter; Vlassopoulos, Dimitris; Ziherl, Primož; Žumer, Slobodan. Topology in soft and biological matter. Phys Rep. 2024;1075:1-137.
-
Röder K, Pasquali S. Assessing RNA atomistic force fields via energy landscape explorations in implicit solvent. Biophys Rev. 2024;1-11.
-
Bussi G, Bonomi M, Gkeka P, Sattler M, Al-Hashimi HM, Auffinger P, Duca M, Foricher Y, Incarnato D, Jones AN, Kirmizialtin S, Krepl M, Orozco M, Palermo G, Pasquali S, Salmon L, Schwalbe H, Westhof E, Zacharias M. RNA dynamics from experimental and computational approaches. Structure. 2024 Sep 5;32(9):1281-1287. doi: 10.1016/j.str.2024.07.019. PMID: 39241758.
-
Divina El Hamaoui, Aurore Marchelli, Sophie Gandrille, Alain Stepanian, Franck Lebrin, et al.. Thrombin-mediated cleavage of membrane endoglin: Implications in endothelial dysfunction. 17th Annual Congress of the European Association for Haemophilia and Allied Disorders 2024, Haemophilia, 30 (S1), pp.34-188, 2024, ⟨10.1111/hae.14919⟩. ⟨hal-04460082⟩
- Rapone R, Del Maestro L, Bouyioukos C, Albini S, Cruz-Tapias P, Joliot V, Cosson B, Ait-Si-Ali S. The cytoplasmic fraction of the histone lysine methyltransferase Setdb1 is essential for embryonic stem cells. iScience. 2023 Jul 14;26(8):107386. doi: 10.1016/j.isci.2023.107386. PMID: 37559904; PMCID: PMC10407132.
- Nguyen BVG, Nguyen HHN, Vo TH, Le MT, Tran-Nguyen VK, Vu TT, Nguyen PV. Prevalence and drug susceptibility of clinical Candida species in nasopharyngeal cancer patients in Vietnam. One Health. 2023 Dec 3;18:100659. doi: 10.1016/j.onehlt.2023.100659. PMID: 38179314; PMCID: PMC10761778.
-
Rossi E, Pericacho M, Kauskot A, Gamella-Pozuelo L, Reboul E, Leuci A, Egido-Turrion C, El Hamaoui D, Marchelli A, Fernández FJ, Margaill I, Vega MC, Gaussem P, Pasquali S, Smadja DM, Bachelot-Loza C, Bernabeu C. Soluble endoglin reduces thrombus formation and platelet aggregation via interaction with αIIbβ3 integrin. J Thromb Haemost. 2023 Jul;21(7):1943-1956. doi: 10.1016/j.jtha.2023.03.023. Epub 2023 Mar 28. PMID: 36990159.
-
Ghoula M, Kongmeneck AD, Eid R, Camproux AC, Moroy G. Comparative study of the mutations observed in the SARS-CoV-2 RBD variants of concern and their impact on the interaction with the ACE2 protein. J Phys Chem B. 2023;127(40):8586-8602. doi: 10.1021/acs.jpcb.3c01467.
-
Lejal V, Cerisier N, Rouquié D, Taboureau O. Assessment of drug-induced liver injury through cell morphology and gene expression analysis. Chem Res Toxicol. 2023;36(9):1456-1470. doi: 10.1021/acs.chemrestox.2c00381.
-
Tran-Nguyen VK, Ballester PJ. Beware of simple methods for structure-based virtual screening: the critical importance of broader comparisons. J Chem Inf Model. 2023;63(5):1401-1405. doi: 10.1021/acs.jcim.3c00218.
-
Tran-Nguyen VK, Junaid M, Simeon S, Ballester PJ. A practical guide to machine-learning scoring for structure-based virtual screening. Nat Protoc. 2023 Nov;18(11):3460-3511. doi: 10.1038/s41596-023-00885-w. Epub 2023 Oct 16. PMID: 37845361.
A lire aussi…
Actualités de l’équipe
Le rôle clé de la communication intestin-cerveau dans le circuit de la récompense

La théorie du vieillissement
La théorie du vieillissement par Michaël Reira. Epsiloon le magazine scientifique d’actualité

Un monde sous plastique, enquête sur une crise sanitaire et environnementale au 21è siècle
Retrouvez, via le lien ci dessous, les détails du cycle de 12 heures avec les coordinatrices Francelyne MARANO, Sonja BOLAND et Stéphanie DEVINEAU. Un monde sous plastique, enquête sur une crise sanitaire et environnementale au 21è siècle | Université Ouverte

Du master au doctorat : l’expérience de Nejmeh (C2OFFEE) en Graduate School
Faits marquants
dernière mise à jour 19/02/2025
