Enzymes, environnement, pathologie moléculaire
LC BUI, F BUSI, F DESHAYES, JM DUPRET, E PETIT, F RODRIGUES LIMA, M VIGUIER
Nos recherches portent principalement sur l’acétyltransférase CREBBP, la méthyltransférase SETD2, ainsi que sur les tyrosine phosphatases PTPN2 et PTP1B. Toutefois, dans le cadre de collaborations, nous sommes régulièrement amenés à participer à des travaux sur d’autres systèmes enzymatiques.
CREBBP et SETD2 jouent des rôles majeurs dans les processus épigénétiques, notamment via la modification des histones (en particulier les marques H3K18ac et H3K36me3), mais également par la modification de protéines non histones telles que des facteurs de transcription ou des protéines du cytosquelette.
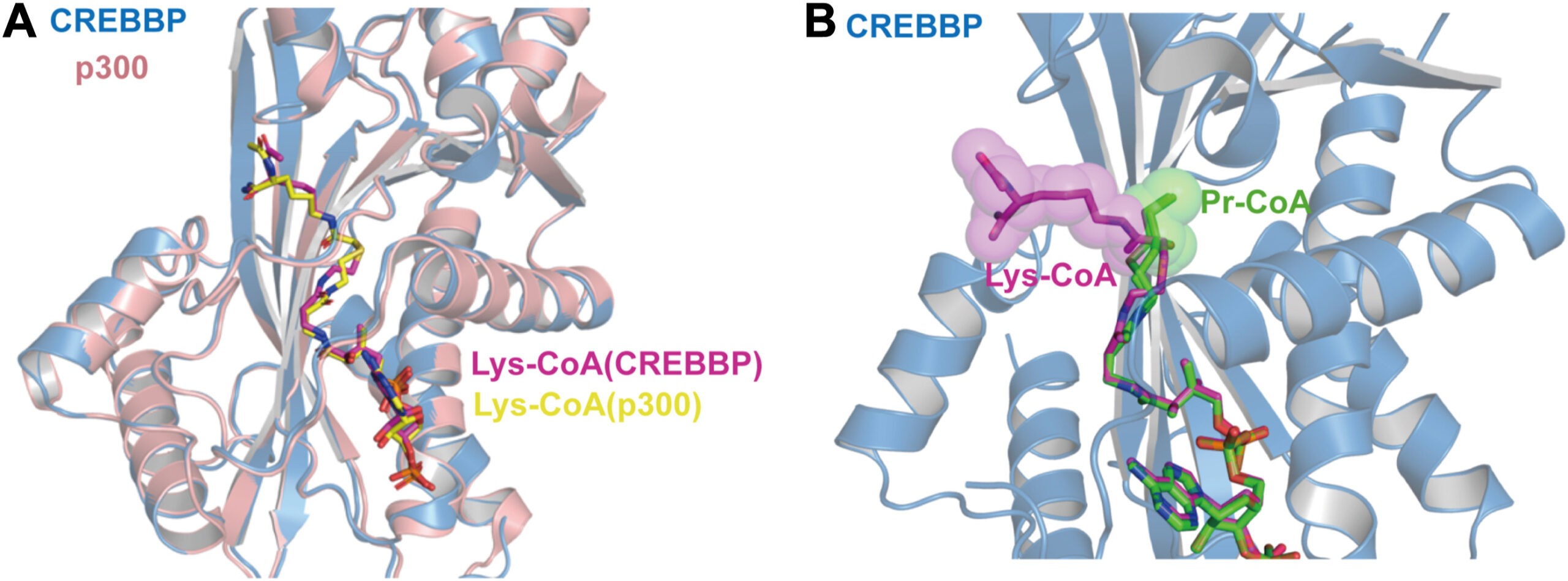
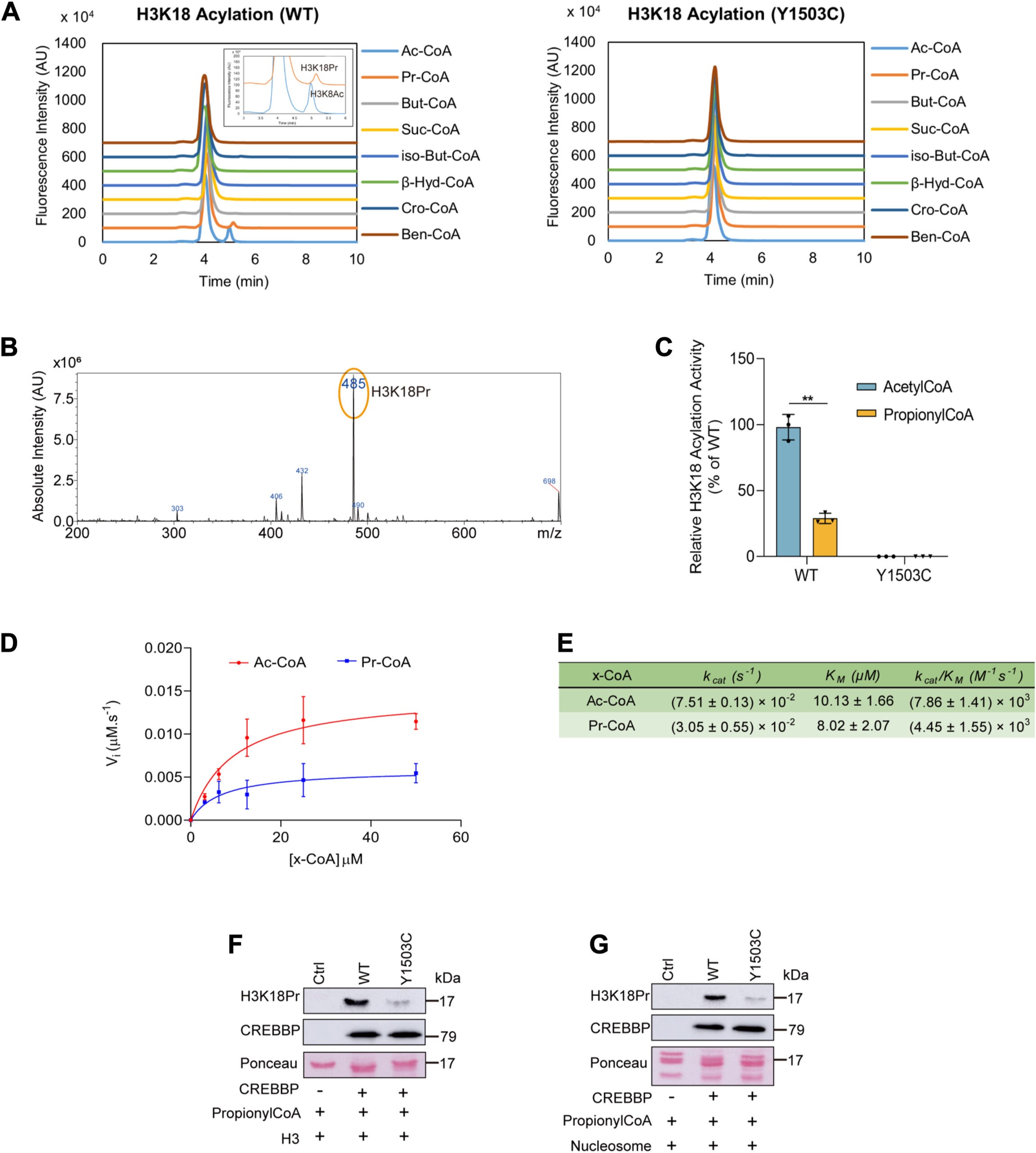
Ces deux enzymes sont considérées comme des suppresseurs de tumeurs, toutefois, leurs implications pathologiques s’étendent au-delà des pathologies tumorales, puisque des dérégulations de CREBBP et de SETD2 sont également impliquées dans des maladies neuro-développementales et métaboliques. À l’aide d’approches moléculaires, cellulaires et structurales, nous avons récemment caractérisé plusieurs formes mutantes de CREBBP et de SETD2 impliquées dans des pathologies hématopoïétiques (Yang et al., Blood, 2025; Michail et al., J. Biol. Chem. 2026). Nous avons par ailleurs démontré que CREBBP possède une activité d’histone propionyltransférase, capable de générer la marque épigénétique H3K18pr, apportant ainsi un éclairage nouveau sur les mécanismes reliant le métabolisme cellulaire aux régulations post-traductionnelles et épigénétiques (Cui et al., J. Biol. Chem. 2025).
Dans le cadre des travaux sur CREBBP et SETD2, nous avons développé plusieurs outils, incluant des dosages enzymatiques spécifiques, des quantifications métaboliques, des modèles cellulaires édités par CRISPR/Cas9, ainsi que des vecteurs permettant l’expression, la purification et la caractérisation structurale de ces enzymes et leurs variants pathologiques.
En parallèle, l’équipe développe depuis plusieurs années un savoir-faire sur certaines tyrosine phosphatases, en particulier PTPN2 et PTP1B. Ces enzymes sont impliquées dans plusieurs voies de signalisation cellulaire, et leur dérégulation est associée à des pathologies tumorales, inflammatoires et métaboliques. Nous avons caractérisé, aux échelles moléculaire, cellulaire et structurale, une forme mutante de PTPN2 responsable d’une pathologie inflammatoire de l’intestin (Parlatto et al., Gastroenterology, 2020; Nian et al., Protein Sci., 2022). Plus récemment, nous avons mis en évidence l’impact de certains composés chimiques (cisplatine utilisé en chimiothérapie et certaines quinones présentes dans des crèmes cosmétiques) sur l’activité de PTP1B et sur les voies de signalisation associées (Liu et al., Biomed. Pharmacother. 2022; Arnaud et al., Mol. Pharmacol. 2025).
De manière similaire aux travaux menés sur CREBBP et SETD2, plusieurs outils ont été développés afin de doser l’activité enzymatique de PTPN2 et de PTP1B, et plus largement d’étudier leur structure et leurs fonctions.
Nos études actuelles sur CREBBP et SETD2 visent à approfondir la compréhension de la structure et des fonctions de ces enzymes, notamment par l’analyse de nouvelles mutations associées à des pathologies neuro-développementales et tumorales. Ces travaux ont pour objectif, d’une part, de caractériser l’impact de ces mutations pathogènes et, d’autre part, d’améliorer notre compréhension de ces enzymes, notamment par l’identification de nouveaux substrats protéiques, de fonctions nouvelles ou d’interactions métaboliques en lien avec le coenzyme A et la S-adénosylméthionine.
Nos travaux actuels sur PTPN2 portent sur la caractérisation de formes mutantes identifiées dans des cancers colorectaux et dans des maladies métaboliques. Nous étudions également l’impact de certains cancérogènes chimiques (cadmium) sur la signalisation dépendante de PTPN2 (notamment dans l’intestin).
Ces travaux s’appuient sur des collaborations nationales et internationales. Par ailleurs, nos recherches bénéficient du plateau technique Bioprofiler de l’Unité et contribuent à son développement, notamment par la mise au point de dosages enzymatiques, de détection et caractérisation de molécules/métabolites.